利用Databricks Delta大规模简化基因组学管道
2019年3月7日 在工程的博客
在数据库试试这个笔记本
这篇博客是我们“大规模基因组学分析”系列的第一篇博客。在本系列中,我们将演示如何Databricks基bob体育亚洲版因组学统一分析平台bob体育客户端下载使客户能够分析人口规模的基因组数据。从我们的输出开始基因组学管道,本系列将提供使用Databricks运行样本质量控制、联合基因分型、队列质量控制和高级统计遗传学分析的教程。
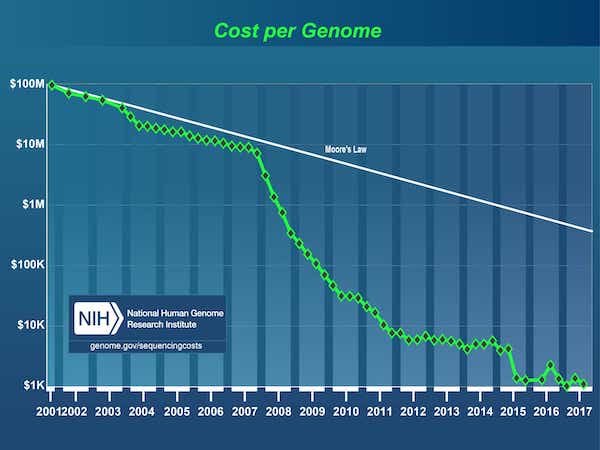
自建成以来人类基因组计划2003年,DNA测序的成本从30亿美元大幅下降,这推动了数据的爆炸式增长1第一个基因组的价格在今天低于1000美元。
[1]人类基因组计划是一项耗资30亿美元的项目,由能源部和国立卫生研究院(National Institutes of Health)牵头,始于1990年,于2003年完成。
来源:DNA测序成本:数据
因此,基因组学领域现在已经成熟到一个阶段,公司已经开始在人口规模上进行DNA测序。然而,对DNA编码进行测序只是第一步,然后需要将原始数据转换为适合分析的格式。通常这是通过粘合在一起的一系列生物信息学带有自定义脚本的工具,并在单个节点上处理数据,每次一个样本,直到我们得到基因组变体的集合。生物信息学今天的科学家大部分时间都花在建造和维护这些管道上。随着基因组数据集扩展到pb级,及时回答以下简单问题也变得具有挑战性:
- 这个月我们测序了多少样品?
- 检测到的唯一变异的总数是多少?
- 在不同种类的变异中我们看到了多少变异?
使这个问题进一步复杂化的是,来自成千上万个人的数据在保持可访问和可查询的同时,无法存储、跟踪或版本化。因此,研究人员在进行分析时经常复制基因组数据的子集,导致整体存储空间和成本上升。为了缓解这一问题,如今的研究人员采用了一种“数据冻结”策略,通常是在6个月到2年之间,他们停止对新数据的研究,而是专注于现有数据的冻结副本。没有办法在更短的时间内逐步建立分析,导致研究进展放缓。
人们迫切需要一种强大的软件,既能在工业规模上消费基因组数据,又能保持科学家探索数据、迭代分析管道并获得新见解的灵活性。
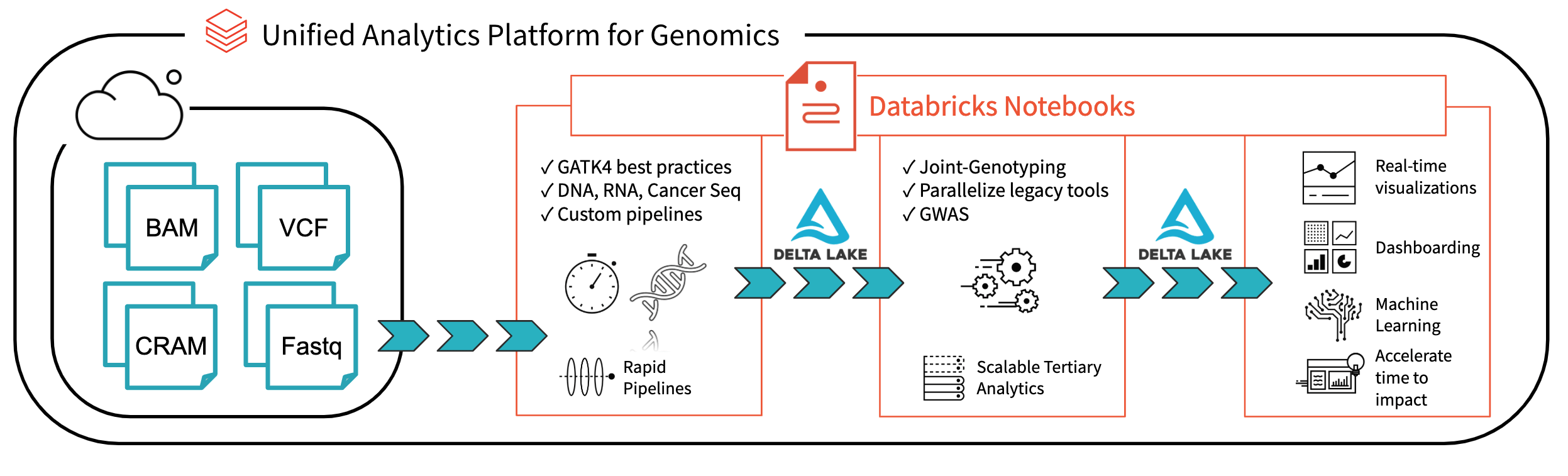
图1所示。使用Databricks进行端到端基因组学分析的体系结构
与Databricks Delta:实时大数据分析的统一管理系统, Databricks平台在解决bob体育客户端下载当今研究人员面临的数据治理、数据访问和数据分析问题方面迈出了重要一步。与三角洲湖在美国,你可以将所有的基因组数据存储在一个地方,并创建分析,在摄入新数据时实时更新。结合我们的优化bob体育亚洲版基因组学统一分析平台bob体育客户端下载(UAP4G)用于读取、写入和处理基因组文件格式,我们为基因组管道工作流程提供端到端解决方案。UAP4G架构提供了灵活性,允许客户插入自己的管道并开发自己的三级分析。例如,我们突出显示了以下显示质量控制指标和可视化的仪表板,这些指标和可视化可以以自动的方式计算和显示,并根据您的特定需求进行定制。
https://www.youtube.com/watch?v=73fMhDKXykU
在本博客的其余部分中,我们将详细介绍构建上面的质量控制仪表板的步骤,该仪表板在示例完成处理时实时更新。通过使用基于delta的管道来处理基因组数据,我们的客户现在可以以一种提供实时、逐个样本可见性的方式操作他们的管道。通过Databricks笔记本(以及GitHub和MLflow等集成),他们可以跟踪和版本分析,以确保结果的可重复性。他们的生物信息学家可以花更少的时间来维护管道,而花更多的时间来进行发现。我们将UAP4G视为引擎,将推动从特别分析到工业规模生产基因组学的转变,使人们能够更好地了解遗传和疾病之间的联系。
读取样本数据
让我们先从一小群样本中读取变异数据;下面的语句读入特定sampleId的数据,并使用Databricks Delta格式保存它(在delta_stream_output文件夹中)。
spark.read。\格式(“铺”).\负载(“dbfs: / annotations_etl_parquet sampleId = "+“SRS000030_SRR709972”).\写。\格式(“δ”).\保存(delta_stream_outpath)注意,annotations_etl_parquet文件夹包含从1000个基因组数据集存储在拼花格式。这些注释的ETL和处理是使用Databricks的基bob体育亚洲版因组学统一分析平台bob体育客户端下载.
开始流数据Delta表
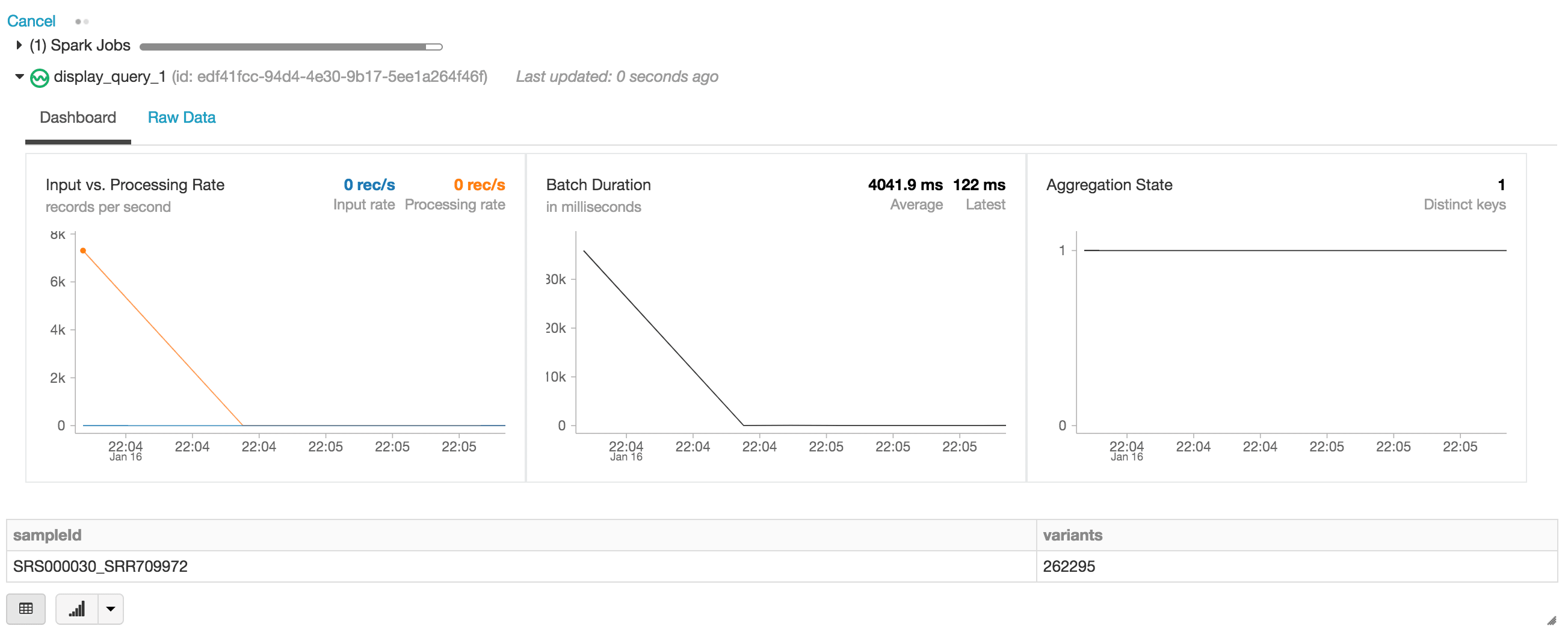
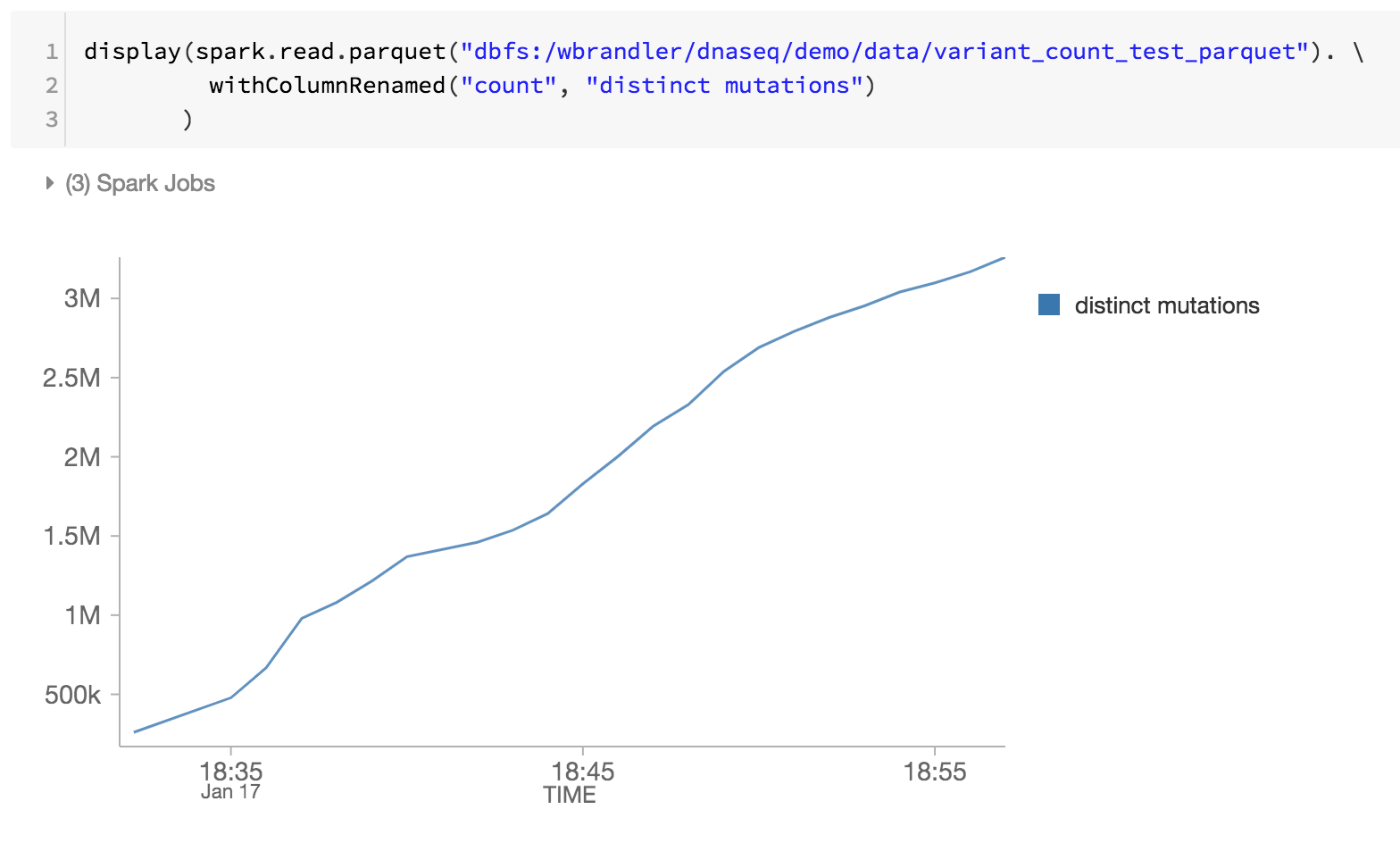
在下面的语句中,我们正在创建exomes Apache Spark DataFrame,它正在使用Databricks Delta格式读取数据流(通过readStream)。这是一个持续运行或动态的DataFrame,也就是说,当数据被写入delta_stream_output文件夹时,exomes DataFrame将加载新的数据。要查看外显子组的DataFrame,我们可以运行一个DataFrame查询来查找根据sampleId分组的变体的计数。
#读取数据流exomes = spark.readStream。格式(“δ”) .load (delta_stream_outpath)通过DataFrame查询显示数据显示器(exomes.groupBy (“sampleId”) .count () .withColumnRenamed (“数”,“变异”))当执行显示语句,Databricks笔记本提供了一个流仪表板来监视流作业。紧接在流作业下面的是display语句的结果(即sample_id变量的计数)。
让我们继续回答我们最初的一组问题,通过运行基于我们的DataFrame的其他查询外显DataFrame。
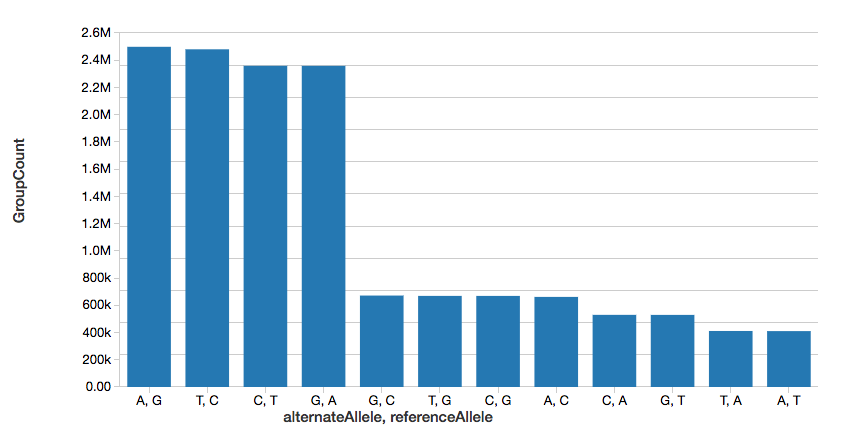
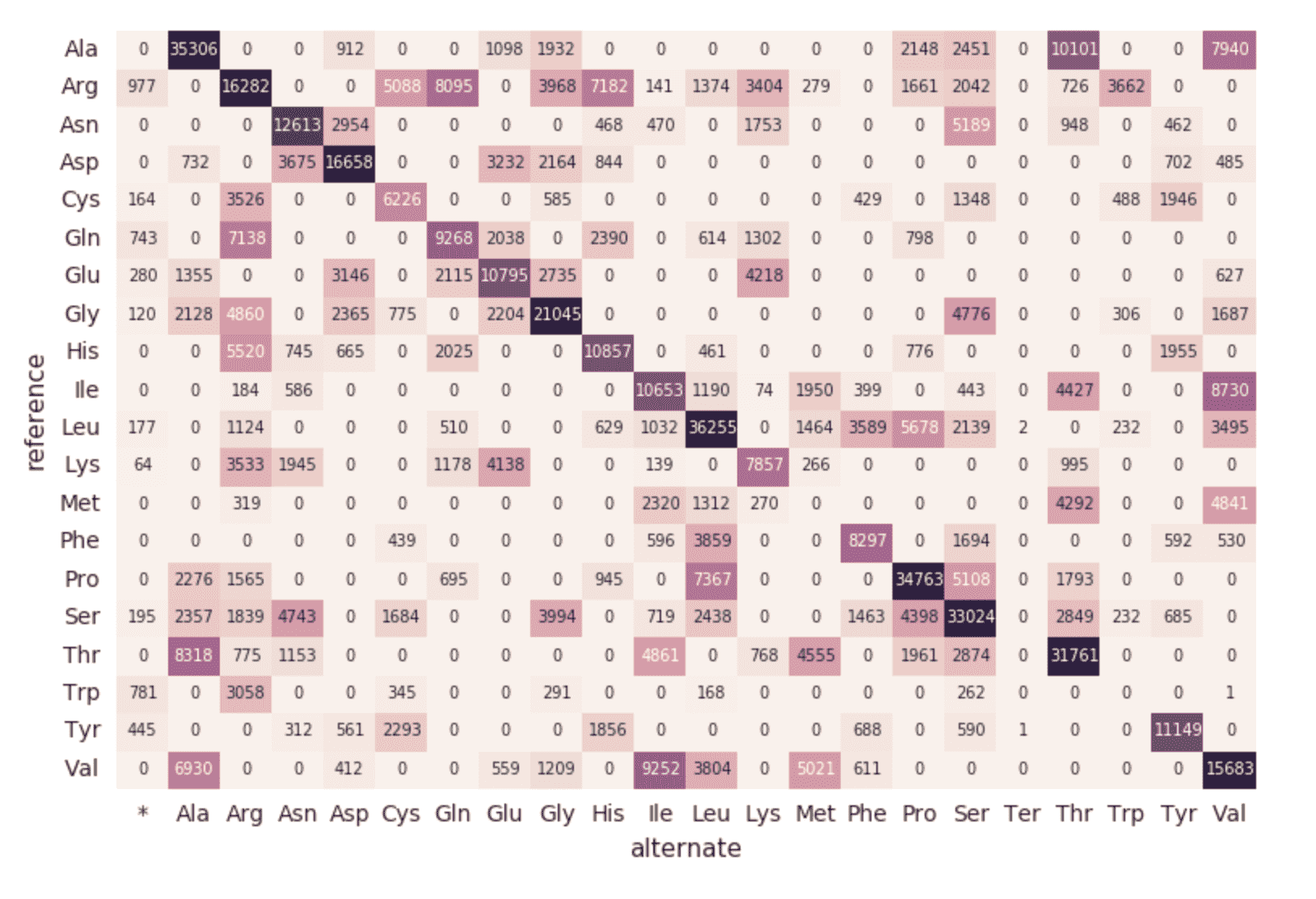
单核苷酸变异数
为了继续这个例子,我们可以快速计算出单核苷酸变体(SNVs)的数量,如下图所示。
%sql选择referenceAllele alternateAllele,数(1)作为GroupCount从snvs集团通过referenceAllele, alternateAllele订单通过GroupCountdesc注意,display命令是Databricks工作空间的一部分,它允许您使用Databricks可视化(即不需要编码)查看您的DataFrame。
变异数
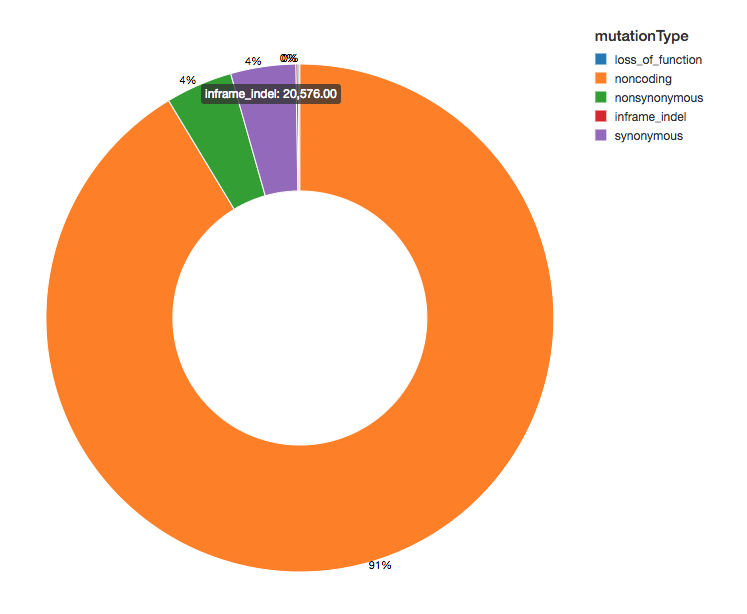
既然我们已经用功能效应注解了我们的变体,我们可以通过观察我们看到的变体效应的扩散来继续我们的分析。大多数变体检测到的蛋白质编码区域,这些被称为非编码变体。
显示器(exomes.groupBy(“mutationType”)。数())氨基酸替代热图
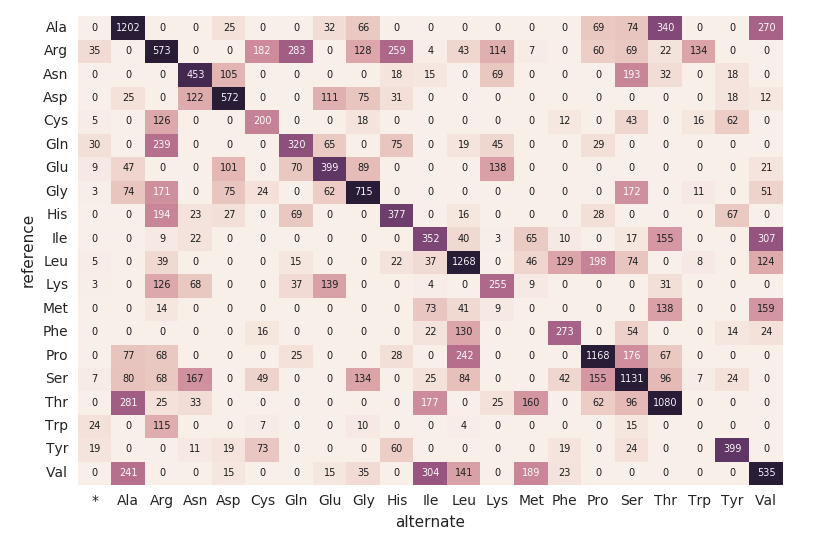
继续我们的外显DataFrame,让我们用下面的代码片段计算氨基酸替代计数。类似于前面的数据帧,我们将创建另一个动态数据帧(aa_counts)因此,当新数据被外显子组DataFrame处理时,它随后也会反映在氨基酸替代计数中。我们还将数据写入内存(例如:.format(“记忆”)),每60秒处理一批(即触发(processingTime = 60秒)),这样下游的Pandas热图代码就可以处理和可视化热图。
计算氨基酸替换计数编码= get_coding_mutations(外显子组)Aa_substitutions = get_amino_acid_substitutions(coding.select(“proteinHgvs”),“proteinHgvs”)Aa_counts = count_aminino_acid_substitution_combination (aa_substitutiontions)aa_counts。\writeStream。\格式(“记忆”).\queryName (“amino_acid_substitutions”).\outputMode (“完整的”).\触发(processingTime =“60秒”).\start ()下面的代码片段读取了前面的内容amino_acid_substitutionsSpark表,确定最大计数,从Spark表创建一个新的Pandas pivot表,然后绘制热图。
#使用熊猫而且matplotlib来构建的热图amino_acid_substitutions=spark.read.table(“amino_acid_substitutions”)max_count=amino_acid_substitutions.agg(外汇。马克斯(“替换”))。收集() (0] [0]aa_counts_pd=amino_acid_substitutions.toPandas ()aa_counts_pd=pd.pivot_table (aa_counts_pd值=“替换”、索引=[“参考”),列=[“替代”), fill_value=0)无花果、斧头=plt.subplots ()与sns.axes_style(“白”):斧头=sns。热图(aa_counts_pd,=max_count*0.4, cbar=假, annot=真正的, annot_kws={“大小”:7}, fmt=“d”)plt.tight_layout ()显示(图)迁移到一个连续的管道
到目前为止,前面的代码片段和可视化表示对单个运行的单个运行sampleId.但是因为我们使用Structured Streaming和Databricks Delta,所以可以使用这段代码(无需任何更改)构建一个生产数据管道,当样本在管道中滚动时,它可以连续计算质量控制统计数据。为了演示这一点,我们可以运行下面的代码片段,它将加载整个数据集。
进口时间拼花=“dbfs: / databricks-datasets /基因组学/ annotations_etl_parquet /”Files = dbutils.fs.ls(parquets)counter =0为样本在文件:计数器+ =1Annotation_path = sample.pathsampleId =注释路径。split(“/”) [4] .split (“=”) [1]变体= spark.read。格式(“铺”) .load (str(annotation_path))打印(“运行”+ sampleId)如果(sampleId ! =“SRS000030_SRR709972”):variants.write。格式(“δ”).\模式(“添加”).\保存(delta_stream_outpath)time . sleep (10)的源代码,如前面的代码片段所述外显DataFrame是加载到delta_stream_output文件夹中。最初,我们为单个文件加载了一组文件sampleId(例如,sampleId = " SRS000030_SRR709972 ").上面的代码片段现在使用所有生成的拼花示例(即。拼花)和增量加载这些文件sampleId变成相同的delta_stream_output文件夹中。下面的GIF动画显示了前面代码段的简短输出。
https://www.youtube.com/watch?v=JPngSC5Md-Q
可视化您的基因组学管道
当您滚动回笔记本的顶部时,您将注意到外显DataFrame现在自动加载新的sampleIds.因为基因组学管道的结构化流组件持续运行,所以一旦新文件加载到delta_stream_outputpath文件夹中。通过使用Databricks Delta格式,我们可以确保进入exomes DataFrame的数据流的事务一致性。
https://www.youtube.com/watch?v=Q7KdPsc5mbY
而不是我们最初的创造外显DataFrame,请注意结构化流监控仪表板现在是如何加载数据的(即,波动的“输入与处理速率”、波动的“批处理持续时间”以及“聚合状态”中不同键的增加)。随着外显DataFrame正在处理,请注意新的行sampleIds(以及变量计数)。同样的操作也可以在关联的对象中看到按突变类型分组查询.
https://www.youtube.com/watch?v=sT179SCknGM
使用Databricks Delta,任何新数据在我们基因组管道的每一步都是事务一致的。这很重要,因为它确保了管道的一致性(维护数据的一致性,即确保所有数据都是“正确的”)、可靠性(事务成功或完全失败),并且可以处理实时更新(同时处理许多事务的能力,任何故障中断都不会影响数据)。因此,即使是我们下游氨基酸替代图中的数据(其中有许多额外的ETL步骤)也会无缝刷新。
作为基因组学管道的最后一步,我们还通过检查DBFS中的Databricks Delta parquet文件来监控不同的突变(即随着时间的推移,不同突变的增加)。
总结
使用Databricks统一分析平台的基础-特别是Databricks Delbob体育亚洲版ta -生物信息学家bob体育客户端下载和研究人员可以应用具有事务一致性的分布式分析Databricks基bob体育亚洲版因组学统一分析平台bob体育客户端下载.这些抽象允许数据从业者简化基因组学管道。在这里,我们创建了一个基因组样本质量控制管道,随着新样本的处理不断处理数据,无需人工干预。无论您是执行ETL还是执行复杂的分析,您的数据都将通过基因组学管道快速流动,而不会中断。自己试试吧,今天就下载用Databricks Delta笔记本电脑大规模简化基因组学管道.
开始大规模分析基因组学:
- 阅读我们的基因组学bob体育亚洲版统一分析解决方案指南
- 下载用Databricks Delta笔记本电脑大规模简化基因组学管道
- 报名参加免费试用Databricks基因组学bob体育亚洲版统一分析公司
致谢
感谢黄永胜和Michael Ortega的贡献。
参观三角洲湖在线中心要了解BOB低频彩更多,请下载最新代码并加入Delta Lake社区。